Tradução do RNA: o que é, como ocorre
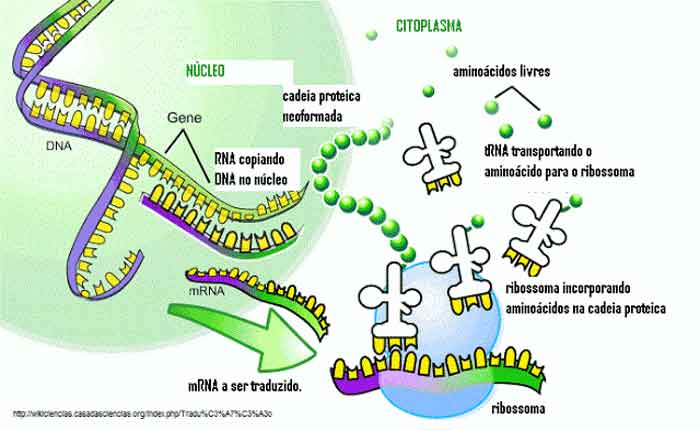
A síntese de proteínas é um dos processos metabólicos mais consumidores de energia de uma célula. Por sua vez, as proteínas respondem por mais massa do que qualquer outro componente dos organismos vivos (com exceção da água), e as proteínas desempenham uma ampla variedade de funções de uma célula. O processo de tradução, ou síntese proteica, envolve a descodificação de uma mensagem de ARNm num produto polipeptídico. Os aminoácidos são ligados covalentemente em comprimentos que variam de aproximadamente 50 aminoácidos a mais de 1000.
A maquinaria de síntese de proteínas
Além do modelo de RNAm, muitas outras moléculas contribuem para o processo de tradução. A composição de cada componente pode variar entre as espécies; por exemplo, os ribossomos podem consistir em números diferentes de RNAs ribossômicos ( rRNA ) e polipeptídeos dependendo do organismo. No entanto, as estruturas gerais e funções da maquinaria de síntese de proteínas são comparáveis de bactérias para células humanas. A tradução requer a entrada de um modelo de RNAm, ribossomos, RNAt e vários fatores enzimáticos ( Figura ).

Em E. coli , existem 200.000 ribossomos presentes em todas as células em um dado momento. Um ribossomo é uma macromolécula complexa composta de rRNAs estruturais e catalíticos e muitos polipeptídeos distintos. Em eucariotos, o nucléolo é completamente especializado para a síntese e montagem de rRNAs.
Os ribossomos estão localizados no citoplasma em procariontes e no citoplasma e retículo endoplasmático de eucariotos. Os ribossomos são compostos de uma subunidade grande e uma pequena que se juntam para a tradução.
A subunidade pequena é responsável pela ligação do molde de ARNm, enquanto a subunidade grande se liga sequencialmente aos ARNt , um tipo de molécula de ARN que traz aminoácidos para a cadeia de crescimento do polipeptídeo. Cada molécula de mRNA é traduzida simultaneamente por muitos ribossomos, todos sintetizando proteínas na mesma direção.
Dependendo da espécie, existem 40 a 60 tipos de RNAt no citoplasma. Servindo como adaptadores, os ARNt específicos ligam-se a sequências no molde de ARNm e adicionam o aminoácido correspondente à cadeia polipeptídica. Portanto, tRNAs são as moléculas que realmente “traduzem” a linguagem do RNA para a linguagem das proteínas. Para cada ARNt funcionar, ele deve ter seu aminoácido específico ligado a ele. No processo de carregamento de tRNA, cada molécula de tRNA é ligada ao seu aminoácido correto.
O código genético
Para resumir o que sabemos até agora, o processo celular de transcrição gera RNA mensageiro (mRNA), uma cópia molecular móvel de um ou mais genes com um alfabeto de A, C, G e uracil (U). A tradução do modelo de RNAm converte informações genéticas baseadas em nucleotídeos em um produto proteico.
Sequências de proteínas consistem em 20 aminoácidos que ocorrem com frequência; portanto, pode-se dizer que o alfabeto proteico consiste em 20 letras. Cada aminoácido é definido por uma sequência de três nucleotídeos chamada de códon tripleto . A relação entre um codão nucleotídico e o seu correspondente aminoácido é designada por código genético .
Dados os números diferentes de “letras” no mRNA e nos “alfabetos” de proteínas, combinações de nucleotídeos correspondiam a aminoácidos isolados. Usando um código de três nucleotídeos significa que há um total de 64 (4 × 4 × 4) combinações possíveis; portanto, um determinado aminoácido é codificado por mais de um tripleto de nucleotídeos ( Figura ).

Três dos 64 codões terminam a síntese proteica e libertam o polipéptido da maquinaria de tradução. Estes trigêmeos são chamados de códons de parada . Outro codon, AUG, também tem uma função especial. Além de especificar o aminoácido metionina, ele também serve como o códon inicial para iniciar a tradução.
O quadro de leitura para tradução é definido pelo codão de início AUG próximo da extremidade 5 ‘do mRNA. O código genético é universal. Com poucas exceções, praticamente todas as espécies usam o mesmo código genético para a síntese de proteínas, o que é uma forte evidência de que toda a vida na Terra compartilha uma origem comum.
Para entender melhor esse assunto veja também:
- Replicação do DNA: entendas as etapas de Duplicação do DNA
- Organização e Estrutura do DNA
- O que é DNA polimerase: definição, como funciona
- O que é DNA recombinante: como funciona
- Transcrição do DNA: o que é, como acontece, pra que serve
- Transcrição do DNA: o que é, como acontece, pra que serve
- Tradução do RNA: o que é, como ocorre
- Tradução do RNA: o que é, como ocorre
O Mecanismo da Síntese Proteica
Assim como na síntese de mRNA, a síntese de proteínas pode ser dividida em três fases: iniciação, alongamento e terminação. O processo de tradução é semelhante em procariotas e eucariotas. Aqui, vamos explorar como a tradução ocorre em E. coli , um procariota representativo, e especificar quaisquer diferenças entre a tradução procariótica e eucariótica.
A síntese proteica começa com a formação de um complexo de iniciação. Em E. coli , esse complexo envolve a subunidade ribossômica pequena, o modelo de RNAm, três fatores de iniciação e um RNAt iniciador especial. O ARNt iniciador interage com o codão de início AUG e liga-se a uma forma especial do aminoácido metionina que é tipicamente removida do polipéptido após a tradução estar completa.
Em procariotas e eucariotos, os fundamentos do alongamento do polipeptídeo são os mesmos, então vamos rever o alongamento da perspectiva da E. coli . A grande subunidade ribossômica de E. coliconsiste em três compartimentos: o sítio A liga os tRNAs carregados de entrada (tRNAs com seus aminoácidos específicos anexados).
O local P liga os ARNt carregados portadores de aminoácidos que formaram ligações com a cadeia polipeptídica em crescimento, mas que ainda não se dissociaram do seu ARNt correspondente. O local E libera tRNAs dissociados para que possam ser recarregados com aminoácidos livres. O ribossomo muda um códon por vez, catalisando cada processo que ocorre nos três locais. Com cada passo, um ARNt carregado entra no complexo, o polipéptido torna-se um aminoácido por mais tempo e um ARNt não carregado afasta-se. A energia para cada ligação entre os aminoácidos é derivada do GTP, uma molécula semelhante ao ATP ( Figura ). Surpreendentemente, a E. coliO aparelho de tradução demora apenas 0,05 segundos para adicionar cada aminoácido, o que significa que um polipéptido de 200 aminoácidos pode ser traduzido em apenas 10 segundos.

A terminação da tradução ocorre quando um codon de parada (UAA, UAG ou UGA) é encontrado. Quando o ribossoma encontra o codão de terminação, o polipéptido em crescimento é libertado e as subunidades do ribossoma dissociam-se e deixam o ARNm. Depois de muitos ribossomos terem completado a tradução, o mRNA é degradado para que os nucleotídeos possam ser reutilizados em outra reação de transcrição.
Resumo
O dogma central descreve o fluxo de informação genética na célula de genes para mRNA para proteínas. Os genes são usados para fazer mRNA pelo processo de transcrição; O mRNA é usado para sintetizar proteínas pelo processo de tradução. O código genético é a correspondência entre o codon mRNA de três nucleotídeos e um aminoácido.
O código genético é “traduzido” pelas moléculas de tRNA, que associam um códon específico a um aminoácido específico. O código genético é degenerado porque 64 códons tripletos no mRNA especificam apenas 20 aminoácidos e três códons de parada. Isso significa que mais de um códon corresponde a um aminoácido. Quase todas as espécies do planeta usam o mesmo código genético.
Os atores em tradução incluem o modelo de RNAm, ribossomos, RNAt e vários fatores enzimáticos. A pequena subunidade ribossomal liga-se ao modelo de ARNm. A tradução começa no início do AUG no mRNA. A formação de ligações ocorre entre aminoácidos sequenciais especificados pelo modelo de RNAm de acordo com o código genético.
O ribossomo aceita tRNAs carregados e, à medida que avança ao longo do mRNA, catalisa a ligação entre o novo aminoácido e o final do polipeptídeo em crescimento. Todo o mRNA é traduzido em “etapas” de três nucleotídeos do ribossomo. Quando um códon de parada é encontrado, um fator de liberação liga e dissocia os componentes e libera a nova proteína.
Referências:
- https://www.nature.com/scitable/definition/translation-rna-translation-173
- https://www.nature.com/scitable/topicpage/translation-dna-to-mrna-to-protein-393
- http://www.open.edu/openlearn/science-maths-technology/science/biology/what-do-genes-do/content-section-5
Glossário
- códon
- três nucleotídeos consecutivos em ARNm que especificam a adição de um aminoácido especifico d
- Código genético
- os aminoácidos que correspondem aos codões de três nucleotídeos do mRNA
- rRNA
- RNA ribossômico; moléculas de RNA que se combinam para formar parte do ribossomo
- parar o códon
- um dos três códons mRNA que especifica a terminação da tradução
- iniciar códon
- o AUG (ou, raramente, GUG) em um mRNA do qual a tradução começa; sempre especifica metionina
- ARNt
- transferir RNA; uma molécula de RNA que contém uma sequência anticódon específica de três nucleotídeos para emparelhar com o códon de mRNA e também se liga a um aminoácido específico