Replicação do DNA: entendas as etapas de Duplicação do DNA
Quando uma célula se divide, é importante que cada célula filha receba uma cópia idêntica do DNA. Isso é realizado pelo processo de replicação do DNA. A replicação do DNA ocorre durante a fase de síntese, ou fase S, do ciclo celular, antes que a célula entre na mitose ou na meiose.
A elucidação da estrutura da dupla hélice forneceu uma sugestão de como o DNA é copiado. Lembre-se de que os nucleotídeos de adenina se combinam com nucleotídeos de timina e citosina com guanina. Isso significa que as duas vertentes são complementares entre si. Por exemplo, uma cadeia de ADN com uma sequência nucleotídica de AGTCATGA terá uma cadeia complementar com a sequência TCAGTACT ( Figura ).

Por causa da complementaridade dos dois filamentos, ter um filamento significa que é possível recriar o outro filamento. Este modelo para replicação sugere que os dois filamentos da dupla hélice se separam durante a replicação, e cada filamento serve como modelo a partir do qual a nova cadeia complementar é copiada ( Figura ).

Durante a replicação do DNA, cada um dos dois filamentos que compõem a dupla hélice serve como um modelo a partir do qual novas cadeias são copiadas. A nova vertente será complementar à vertente parental ou “antiga”. Cada novo cordão duplo consiste em um filamento parental e um novo filamento filiado. Isso é conhecido como replicação semi conservativa . Quando duas cópias de DNA são formadas, elas têm uma seqüência idêntica de bases nucleotídicas e são divididas igualmente em duas células-filhas.
Replicação de DNA em eucariotos
Como os genomas eucarióticos são muito complexos, a replicação do DNA é um processo muito complicado que envolve várias enzimas e outras proteínas. Ocorre em três etapas principais: iniciação, alongamento e terminação.
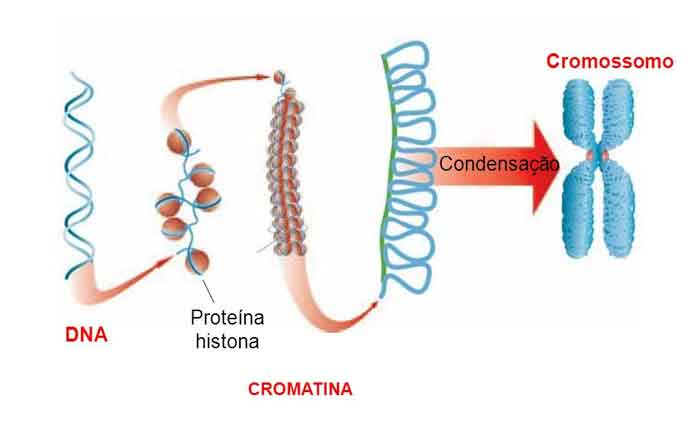
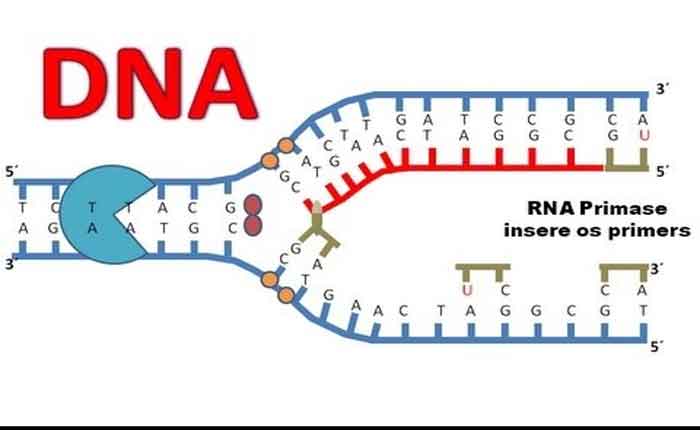
Lembre-se que o DNA eucariótico está ligado a proteínas conhecidas como histonas para formar estruturas chamadas nucleossomas. Durante a iniciação, o DNA é disponibilizado para as proteínas e enzimas envolvidas no processo de replicação. Como a maquinaria de replicação sabe onde a dupla hélice do DNA deve começar? Acontece que existem sequências específicas de nucleotídeos chamadas origens de replicação nas quais a replicação começa. Certas proteínas ligam-se à origem da replicação, enquanto uma enzima chamada helicase desenrola e abre a hélice do DNA. À medida que o DNA se abre, estruturas em forma de Y chamadas de garfos de replicação são formadas ( Figura). Dois linhas de replicação são formados na origem da replicação, e esses são estendidos em ambas as direções conforme a replicação continua. Existem múltiplas origens de replicação no cromossomo eucariótico, de modo que a replicação pode ocorrer simultaneamente a partir de vários locais no genoma.
Durante o alongamento, uma enzima chamada DNA polimerase adiciona os nucleotídeos do DNA à extremidade 3 ‘do modelo. Como a DNA polimerase só pode adicionar novos nucleotídeos no final de um backbone, uma sequência primer , que fornece este ponto de partida, é adicionada com nucleotídeos de RNA complementares. Este iniciador é removido mais tarde e os nucleótidos são substituídos por nucleótidos de ADN. Uma cadeia, que é complementar à cadeia de DNA parental, é sintetizada continuamente na direção da forquilha de replicação, de modo que a polimerase possa adicionar nucleotídeos nessa direção. Este fio continuamente sintetizado é conhecido como o fio líder . Porque DNA polimerase só pode sintetizar DNA em uma direção de 5 ‘para 3’, a outra nova vertente é montada em pequenos pedaços chamadosFragmentos de Okazaki . Cada fragmento de Okazaki requer um primer feito de RNA para iniciar a síntese. O fio com os fragmentos de Okazaki é conhecido como o fio atrasado . À medida que a síntese prossegue, uma enzima remove o primer RNA, que é então substituído por nucleotídeos de DNA, e os intervalos entre os fragmentos são selados por uma enzima chamada DNA ligase .
O processo de replicação do DNA pode ser resumido da seguinte forma:
- O DNA se desenrola na origem da replicação.
- Novas bases são adicionadas às cadeias parentais complementares. Um novo fio é feito continuamente, enquanto o outro é feito em pedaços.
- Os primers são removidos, novos nucleotídeos de DNA são colocados no lugar dos primers e o backbone é selado pela DNA ligase.

Você isola uma cepa celular na qual a junção de fragmentos de Okazaki é prejudicada e suspeita que uma mutação ocorreu em uma enzima encontrada na bifurcação de replicação. Qual enzima tem maior probabilidade de sofrer mutação?
Para entender melhor esse assunto veja também:
- Replicação do DNA: entendas as etapas de Duplicação do DNA
- Organização e Estrutura do DNA
- O que é DNA polimerase: definição, como funciona
- O que é DNA recombinante: como funciona
- Transcrição do DNA: o que é, como acontece, pra que serve
- Regulação da expressão gênica: como o genes são regulados
- Tradução do RNA: o que é, como ocorre
- https://youtu.be/ywiWenzEhWs
Replicação Telômero
Como os cromossomos eucarióticos são lineares, a replicação do DNA chega ao fim de uma linha em cromossomos eucarióticos. Como você aprendeu, a enzima DNA polimerase pode adicionar nucleotídeos em apenas uma direção. No filamento principal, a síntese continua até o final do cromossomo ser atingido; no entanto, na fita atrasada não há lugar para um primer a ser feito para o fragmento de DNA a ser copiado no final do cromossomo. Isso apresenta um problema para a célula, porque as extremidades permanecem sem pareamento e, com o tempo, essas extremidades ficam progressivamente mais curtas à medida que as células continuam a se dividir. As extremidades dos cromossomos lineares são conhecidas como telômeros, que têm sequências repetitivas que não codificam para um gene particular. Como conseqüência, são os telômeros que são encurtados com cada ciclo de replicação do DNA em vez de genes. Por exemplo, em humanos, uma sequência de seis pares de bases, TTAGGG, é repetida 100 a 1000 vezes. A descoberta da enzima telomerase ( Figura ) ajudou na compreensão de como as extremidades dos cromossomos são mantidas. A telomerase se liga ao final do cromossomo, e bases complementares ao modelo de RNA são adicionadas na extremidade da fita de DNA. Uma vez que o molde da fita de retardamento é suficientemente alongado, a DNA polimerase pode agora adicionar nucleotídeos que são complementares às extremidades dos cromossomos. Assim, as extremidades dos cromossomos são replicadas.

A telomerase é tipicamente encontrada ativa em células germinativas, células-tronco adultas e algumas células cancerígenas. Por sua descoberta da telomerase e sua ação, Elizabeth Blackburn ( Figura ) recebeu o Prêmio Nobel de Medicina e Fisiologia em 2009.

A telomerase não é ativa em células somáticas adultas. Células somáticas adultas que sofrem divisão celular continuam a ter seus telômeros encurtados. Isso significa essencialmente que o encurtamento de telômeros está associado ao envelhecimento. Em 2010, os cientistas descobriram que a telomerase pode reverter algumas condições relacionadas à idade em camundongos, e isso pode ter potencial na medicina regenerativa. 1 ratos deficientes em telomerase foram utilizados nestes estudos; esses camundongos têm atrofia tecidual, depleção de células-tronco, falência do sistema de órgãos e respostas prejudicadas a lesões teciduais. A reativação da telomerase nesses camundongos causou a extensão dos telômeros, reduziu o dano ao DNA, reverteu a neuro degeneração e melhorou o funcionamento dos testículos, baço e intestinos. Assim, a reativação de telômeros pode ter potencial para tratar doenças relacionadas à idade em humanos.
Replicação de DNA em procariotas
Lembre-se de que o cromossomo procariótico é uma molécula circular com uma estrutura de enrolamento menos extensa que os cromossomos eucarióticos. O cromossomo eucariótico é linear e altamente enrolado em torno das proteínas. Embora existam muitas semelhanças no processo de replicação do DNA, essas diferenças estruturais exigem algumas diferenças no processo de replicação do DNA nessas duas formas de vida.
A replicação do DNA tem sido extremamente bem estudada em procariontes, principalmente devido ao pequeno tamanho do genoma e ao grande número de variantes disponíveis. Escherichia coli tem 4,6 milhões de pares de bases em um único cromossomo circular, e tudo isso é replicado em aproximadamente 42 minutos, partindo de uma única origem de replicação e continuando ao redor do cromossomo em ambas as direções. Isso significa que aproximadamente 1000 nucleotídeos são adicionados por segundo. O processo é muito mais rápido do que nos eucariotos. A tabela resume as diferenças entre as replicações procariótica e eucariótica.
| Diferenças entre replicações eucariótica e procariótica | ||
|---|---|---|
| Propriedade | Procariontes | Eucariotos |
| Origem de replicação | solteiro | Múltiplo |
| Taxa de replicação | 1000 nucleotídeos / s | 50 a 100 nucleotídeos / s |
| Estrutura cromossômica | circular | linear |
| Telomerase | Não presente | Presente |
Reparo de DNA
DNA polimerase pode cometer erros ao adicionar nucleotídeos. Ele edita o DNA, revisando cada nova base adicionada. Bases incorretas são removidas e substituídas pela base correta, e então a polimerização continua ( Figura a ). A maioria dos erros é corrigida durante a replicação, embora, quando isso não acontece, o mecanismo de reparo de incompatibilidade seja empregado. As enzimas de reparo incompatíveis reconhecem a base incorretamente incorporada e a eliminam do DNA, substituindo-a pela base correta ( Figura b ). Em ainda outro tipo de reparo, reparo de excisão de nucleotídeos, a cadeia dupla de DNA é desenrolada e separada, as bases incorretas são removidas juntamente com algumas bases na extremidade 5 ‘e 3’, e estas são substituídas copiando a matriz com a ajuda da DNA polimerase ( Figura c ). O reparo por excisão de nucleotídeos é particularmente importante na correção dos dímeros de timina, que são causados principalmente pela luz ultravioleta. Num dímero de timina, dois nucleótidos de timina adjacentes um ao outro numa cadeia estão ligados covalentemente entre si em vez das suas bases complementares. Se o dímero não for removido e reparado, isso levará a uma mutação. Indivíduos com falhas em seus genes de reparo por excisão de nucleotídeos mostram extrema sensibilidade à luz solar e desenvolvem câncer de pele no início da vida.

A maioria dos erros é corrigida; se não forem, podem resultar em uma mutação – definida como uma mudança permanente na sequência do DNA. Mutações em genes de reparo podem levar a sérias conseqüências, como o câncer.
Resumo da seção
O DNA se replica por um método semi conservativo no qual cada uma das duas cadeias de DNA parentais atua como um modelo para o novo DNA a ser sintetizado. Após a replicação, cada DNA tem uma cadeia parental ou “antiga” e uma filha ou “nova” vertente.
A replicação em eucariotos começa em múltiplas origens de replicação, enquanto a replicação em procariontes começa a partir de uma única origem de replicação. O DNA é aberto com enzimas, resultando na formação do garfo de replicação. Primase sintetiza um primer RNA para iniciar a síntese por DNA polimerase, que pode adicionar nucleotídeos em apenas uma direção. Um fio é sintetizado continuamente na direção do garfo de replicação; isso é chamado de cadeia principal. A outra vertente é sintetizada em uma direção distante do garfo de replicação, em pequenos trechos de DNA conhecidos como fragmentos de Okazaki. Este fio é conhecido como o fio atrasado. Uma vez completada a replicação, os iniciadores de ARNs substituídos por nucleotídeos de ADN e o ADN selado com ADN-ligase.
As extremidades dos cromossomos eucarióticos representam um problema, pois a polimerase é incapaz de estendê-los sem um primer. A telomerase, uma enzima com um molde de RNA embutido, estende as extremidades copiando o molde de RNA e estendendo uma extremidade do cromossomo. A DNA polimerase pode então estender o DNA usando o primer. Desta forma, as extremidades dos cromossomos são protegidas. Células têm mecanismos para reparar o DNA quando ele é danificado ou erros são cometidos na replicação. Esses mecanismos incluem reparo incompatível para substituir nucleotídeos que são pareados com uma base não complementar e reparo de excisão de nucleotídeos, que remove as bases que estão danificadas, como os dímeros de timina.
Glossário
- Ligase de DNA
- a enzima que catalisa a união de fragmentos de DNA juntos
- DNA polimerase
- uma enzima que sintetiza uma nova cadeia de DNA complementar a um molde
- helicase
- uma enzima que ajuda a abrir a hélice do DNA durante a replicação do DNA, quebrando as ligações de hidrogênio
- cadeia secundária
- durante a replicação da cadeia de 3 ‘para 5’, a cadeia que é replicada em fragmentos curtos e longe do garfo de replicação
- cadeia principal
- o fio que é sintetizado continuamente na direção de 5 ‘para 3’ que é sintetizado na direção do garfo de replicação
- conserto de incompatibilidade
- uma forma de reparo de DNA na qual nucleotídeos não complementares são reconhecidos, extirpados e substituídos por nucleotídeos corretos
- mutação
- uma variação permanente na sequência nucleotídica de um genoma
- reparação de excisão de nucleotídeos
- uma forma de reparo de DNA na qual a molécula de DNA é desenrolada e separada na região do dano de nucleotídeo, os nucleotídeos danificados são removidos e substituídos por novos nucleotídeos usando a fita complementar, e a fita de DNA é resselada e deixada voltar ao seu complemento
- Fragmentos de Okazaki
- os fragmentos de DNA que são sintetizados em pequenos trechos na cadeia atrasada
- primer
- um pequeno trecho de nucleotídeos de RNA que é necessário para iniciar a replicação e permitir que a DNA polimerase se ligue e inicie a replicação
- garfo de replicação
- a estrutura em forma de Y formada durante o início da replicação
- replicação semi conservativa
- o método usado para replicar o DNA no qual a molécula de cadeia dupla é separada e cada cadeia atua como um molde para uma nova cadeia a ser sintetizada, então as moléculas de DNA resultantes são compostas por uma nova cadeia de nucleotídeos e uma antiga cadeia de nucleotídeos
- telomerase
- uma enzima que contém uma parte catalítica e um molde de RNA embutido; ele funciona para manter os telômeros nas extremidades dos cromossomos
- telômero
- o DNA no final dos cromossomos lineares